2025年1月8日,河北大学杜会龙教授团队在国际权威期刊Molecular Plant(IF5Year=21.4,植物学3/265)上在线发表了题为A gap-free complete genome assembly of oat and OatOmics, a multi-omics database 的论文,公布了全球首个超过10 Gb的T2T(端粒到端粒)无间隙栽培六倍体燕麦的参考基因组,并整合基因组、群体变异组、转录组、表型组、重要农艺性状相关基因等信息建立了全球首个燕麦多组学综合数据库OatOmics,为燕麦功能基因组学等基础研究和分子育种等应用研究提供了全面的共享平台。
燕麦(Avena sativa L.)是全球重要的优质饲草和粮食作物,因其营养价值与广泛用途备受关注。在“大食物观”背景下,燕麦在粮食安全与农业可持续发展中地位显著提升。然而,燕麦基因组研究面临重大挑战,其基因组超大(>10 Gb)、高重复和异源多倍体等特性导致解析难度极高。基于传统技术的组装结果普遍存在断裂与覆盖不足的问题。同时,尽管燕麦种质资源丰富,但群体水平的基因型、表型、多组学数据及数据库缺乏,极大地限制了燕麦功能基因组学研究和分子设计育种的推进。特别是,关键农艺性状的功能基因研究基础薄弱,使得高产、优质、抗病、耐逆等性状的改良难以实现规模化应用。新一代基因组学技术的进步,为燕麦完整基因组解析和高质量参考序列构建提供了新希望,而结合多组学数据的整合分析,有望系统地揭示燕麦关键功能基因及调控机制,加速分燕麦子辅助设计育种的实现。这不仅将推动燕麦在农业体系中的广泛应用,也为应对粮食安全和气候变化挑战提供了重要科学支撑。
栽培燕麦T2T无间隙基因组组装:迈向基因组学的新篇章
研究团队综合运用PacBio HiFi、ONT、Illumina和Hi-C等测序技术,成功构建了栽培燕麦完整的基因组图谱,其大小达10.99 Gb,该组装结果覆盖了所有21个着丝粒和42个端粒区域(图1)。后续质量评估显示,该基因组组装具有高连续性、完整性和准确性。该基因组组装不仅弥补了先前组装的空白,而且为揭示燕麦基因组的“暗物质”区域的进化和功能提供了前所未有的机会。在无间隙燕麦基因组组装基础上,研究者们可以更精确地定位和解析基因组中的功能性基因区域,包括与农艺性状相关的基因,为后续的基因功能研究和育种工作提供了坚实的基础。这一突破性的成果不仅为燕麦基因组学研究开辟了新天地,也为其他类似大规模、复杂基因组的组装提供了宝贵的参考。
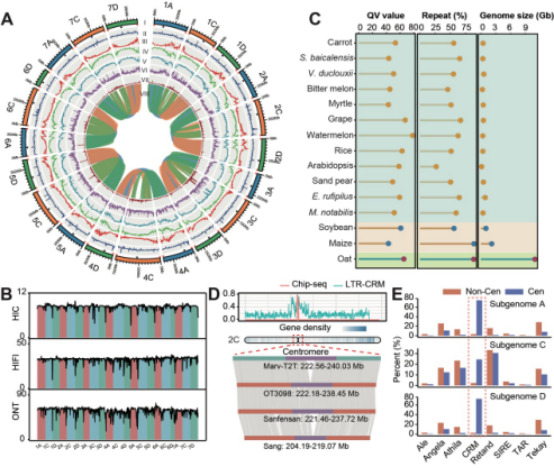
图1 栽培燕麦无间隙基因组组装及质量评估
OatOmics:集成燕麦多组学数据的综合平台
随着组学技术的不断发展,越来越多的基因组、转录组、表型组和遗传变异等多层次的数据被生成并应用于作物改良和育种研究。为了帮助全球科研人员和育种专家高效地利用这些宝贵的资源,杜会龙教授团队开发了OatOmics数据库。OatOmics是一套集成了基因组学、转录组学、基因组变异学和表型组学等多层次数据的综合平台,涵盖了来自全球1,079个燕麦种质资源的变异图谱(图2)。这些种质资源来自不同地区、不同气候环境下的燕麦材料,具有广泛的遗传多样性、表型多样性和适应性,为研究燕麦的遗传改良和表型分析提供了宝贵的参考。
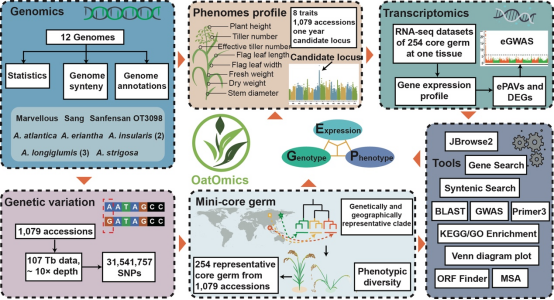
图2 OatOmics数据概览
OatOmics数据库不仅提供了丰富的基因组数据,还涵盖了与农艺性状、抗逆性状、营养成分等相关的转录组数据以及重要的基因变异等信息,同时,OatOmics还集成了基因组浏览器、共线性分析、基因功能富集、序列比对等多组学在线分析工具。这些数据的整合使得科研人员能够从多角度、全方位地探索燕麦的基因组结构、基因功能及其与表型之间的关系。OatOmics数据库的建立,标志着燕麦基因组学研究和分子育种进入了一个全新的时代。作为全球首个集成燕麦多组学数据的数据库,OatOmics为科研人员提供了一个高效、全面的数据平台,推动了燕麦分子育种技术的发展,并为精准改良燕麦品种提供了强有力的支持。
未来,随着燕麦基因组学研究的持续深化,OatOmics数据库将不断扩展和完善,定期更新种质资源、基因变异信息和表型数据,成为全球燕麦研究和育种的核心平台。同时,系统推进种质资源的规模化基因型和表型鉴定,全面掌握资源的全基因组信息、生长特性和生产性状,从中挖掘优异性状和关键基因。随着数据分析和功能注释的持续创新,OatOmics将为分子辅助育种提供更多精准支持,帮助全球燕麦产业应对气候变化、提升生产效益,并满足日益增长的市场需求。OatOmics数据库不仅是燕麦基因组学研究的基础设施,更是推动全球科研人员和育种专家协同合作的桥梁。通过加强国际间的合作和资源共享,OatOmics有望成为燕麦分子育种领域不可或缺的核心工具,助力燕麦品种创新与改良,推动全球粮食安全和可持续农业发展。
河北大学生命科学学院青年教师李伟、科研助理王渝、博士研究生刘佳楠为该论文共同第一作者,河北大学杜会龙教授为论文的通讯作者。定西农科院刘彦明研究员,中国农科院吴斌副研究员,成都大学范昱讲师在种质资源收集等方面提供了重要帮助。极智基因在数据库构建等方面提供了技术支持。河北大学巩志忠教授对本研究给予了指导。该研究得到国家自然科学基金、中国科协青年人才托举工程和河北省自然科学基金等项目的资助。
招聘:河北大学燕麦基因组学与精准设计育种团队因科研工作需要急需基因组学、生物信息学、植物学、遗传学、计算机科学、作物学、基因编辑、遗传育种等方向的优秀博士/博士后/师资博士后/副教授,4-5人!
河北大学燕麦基因组学与精准设计育种团队主要围绕国家重大战略需求,针对我国优异草种资源匮乏和用于饲草产业发展的土地严重不足等问题,拟通过广泛收集全球范围内的野生和栽培燕麦种质资源,利用基因组学、群体遗传学、系统进化生物学和多组学等分析方法,探究燕麦群体内的遗传多样性以及演化历程,揭示燕麦基因组进化与表型可塑性的遗传学基础(生物信息学);进一步结合分子生物学等技术手段挖掘与燕麦产量、抗性等重要农艺性状相关的功能基因,解析燕麦非生物胁迫响应与生长发育平衡的调控网络和分子机制(分子生物学与遗传学);并基于机器学习、基因组预测、基因编辑和杂交等技术实现燕麦的快速从头驯化和定向改造,从而选育高产、优质、耐逆的燕麦新品种(分子设计育种)。