水稻,作为全球最重要的粮食作物之一,对人类的生存和发展起着举足轻重的作用。栽培水稻经长期人工选育,虽在产量与品质等方面展现出显著优势,但遗传多样性的降低使其在应对复杂多变的环境挑战和满足日益增长的粮食需求时深陷困境。野生稻作为栽培稻的近缘物种,它们蕴含着大量有价值的遗传资源,是现代水稻品种遗传改良和种质创新的重要基因宝库。因此,深入挖掘野生稻中蕴含的遗传变异信息,成为突破水稻育种瓶颈、实现可持续发展的关键所在。前期,杜会龙博士利用三代PacBio长片段测序等信息搭建了多个可以显著提高基因组组装质量的流程,成功构建了第一个几乎完整的籼稻R498参考基因组,并通过与包括粳稻日本晴在内的17个水稻基因组进行比较分析,构建了籼粳稻基因组之间较为全面的基因和结构变异图谱,并鉴定到了多个与重要农艺性状相关的结构变异和基因(Du et al., 2017, Nature Communications;Du et al., 2019, Nature Communications)。同时,参与构建了栽培水稻基于图形结构的泛基因组,系统地揭示了水稻中“隐藏”的基因组变异以及其对基因组进化、功能基因表达、水稻驯化和环境适应性等的影响,为水稻功能基因组学和分子设计育种研究提供了重要基础 (Qin et al., 2021, Cell)。

近日,河北大学杜会龙教授团队与江西省农业科学院颜龙安院士团队合作,在国际重要期刊《Nature Communications》(影响因子16.6,中科院一区Top)发表了题为“Genome evolution and diversity of wild and cultivated rice species”的研究论文。该研究通过组装稻属中13个近乎完整的野生稻物种的基因组,并结合已公开的普通野生稻、亚洲栽培稻和非洲栽培稻基因组数据,首次在基因组水平上重构了稻属的进化关系,揭示了各物种的进化轨迹。同时,研究团队构建了目前最为全面的稻属超级泛基因组图谱。利用这些丰富的基因组资源,研究人员进一步挖掘出大量在栽培稻中尚未发现的新基因家族,为水稻的遗传改良和品种创新提供了重要的基础。
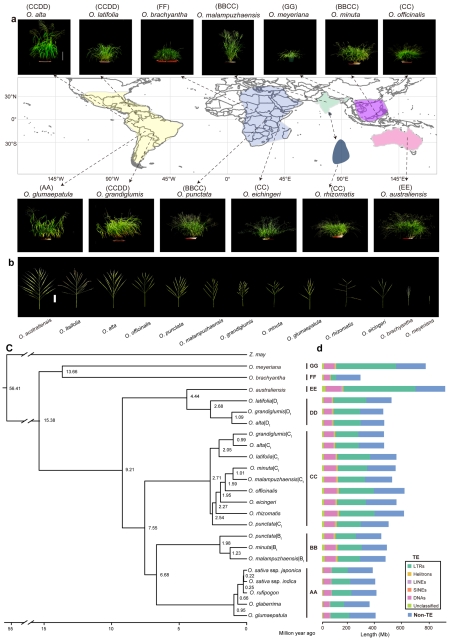
图1. 野生稻与栽培稻的地理分布与系统发育
该研究选择了13种极具代表性的野生稻,其中包含6种异源四倍体和7种二倍体类型。使用PacBio HiFi测序技术和Hi-C测序技术,研究人员成功实现了染色体水平的基因组组装。这些野生稻在地理分布上跨度广泛,表型特征丰富多样,基因组大小显著差异,展现出高度的复杂性和多样性。基于这些高质量的基因组数据,研究人员成功重构了稻属的进化谱系,揭示了稻属物种的遗传演变轨迹。这一发现为深入研究水稻的起源和演化提供新的视角,有助于理解水稻的驯化过程、物种形成机制以及适应环境变化的策略。
通过整合已公开的普通野生稻、亚洲栽培稻以及非洲栽培稻基因组数据,研究团队成功构建了目前最为全面的稻属超级泛基因组,其涵盖了多达101723个基因家族。其中,稻属所共有的核心基因家族占比达9.84%,重要的是,该研究新发现了63,881个在栽培稻中未曾被发现的基因家族。该超级泛基因组蕴含了大量有价值的基因资源,极大地拓展了水稻遗传改良可利用的基因资源池。
该研究在不同水稻材料间鉴定出了大量的结构变异(SVs),包括2,781-10,656个插入序列2,680-10,419个缺失序列、4-52个易位以及7-22个倒位。研究发现,野生稻中SVs的数量更多且长度普遍更大。此外,等位基因变异分析显示,野生稻在等位基因数量以及多样性方面也显著超越栽培稻。这表明野生稻蕴藏着更为丰富的遗传多样性。这些丰富多样的变异不仅为解析水稻适应复杂环境的机制提供了关键线索,同时也为培育高产、优质且抗逆性强的水稻新品种提供了宝贵资源。
江西省农业科学院龙伟雄博士为论文第一作者,河北大学何强青年研究员、硕士研究生王旖焘为论文的共同第一作者,江西省农业科学院谢红卫研究员,河北大学杜会龙教授、江西省农业科学院蔡耀辉研究员和龙伟雄博士为论文的共同通讯作者。中国工程院院士颜龙安院士指导了该项研究。河北大学杜会龙团队科研助理王渝、已毕业硕士生王美佳和李伟青年研究员等也参与了该研究。该研究得到国家自然科学基金和河北省自然科学基金等项目的资助。
论文信息:
Long, W., He, Q., Wang, Y., Wang, Y., Wang, J., Yuan, Z., Wang, M., Chen, W., Luo, L., Luo, L., Xu, W., Li, Y., Li, W., Yan, L., Cai, Y., Du, H., & Xie, H. (2024). Genome evolution and diversity of wild and cultivated rice species. Nature Communications, 15(1), 9994. https://doi.org/10.1038/s41467-024-54427-3
参考文献:
Du, H., Yu, Y., Ma, Y., Gao, Q., Cao, Y., Chen, Z., Ma, B., Qi, M., Li, Y., Zhao, X., Wang, J., Liu, K., Qin, Peng., Yang, X., Zhu, L., Li, S.*, Liang, C.* 2017. Sequencing and de novo assembly of a near complete indica rice genome. Nature Communications, 8,15324.
Du, H., Liang, C. 2019. Assembly of chromosome-scale contigs by efficiently resolving repetitive sequences with long reads. Nature Communications, 10,5360.
Peng Qin; Hongwei Lu; Huilong Du; Hao Wang; Weilan Chen; Zhuo Chen; Qiang He; Shujun Ou; Hongyu Zhang; Xuanzhao Li; Xiuxiu Li; Yan Li; Yi Liao; Qiang Gao; Bin Tu; Hua Yuan; Bingtian Ma; Yuping Wang; Yangwen Qian; Shijun Fan; Weitao Li; Jing Wang; Min He; Junjie Yin; Ting Li; Ning Jiang; Xuewei Chen; Chengzhi Liang; Shigui Li; Pan-genome analysis of 33 genetically diverse rice accessions reveals hidden genomic variations, Cell, 2021, 184(13): 3542-3558.e16.