燕麦作为全球重要的粮饲兼用作物,在保障粮食安全、健康食品供应和缓解畜牧业饲料短缺方面发挥关键作用。燕麦具有耐干旱、耐盐碱、适应贫瘠环境等特点,是我国边际土地高效开发与利用的先锋作物。同时,河北省也是我国燕麦种植面积最大的省份之一,作为河北省的特色经济作物,其在推动乡村振兴、农民增收、产业升级、健康食品供应、畜牧业发展等方面具有重要作用。
近期,河北大学杜会龙/巩志忠教授团队在燕麦基因组学与设计育种研究领域取得多项突破性进展,极大地促进了燕麦基础与应用研究:完成了全球燕麦种质资源的广泛收集与新质生产力的系统性评价,筛选了120余份高产、优质、耐逆的优异燕麦种质资源,构建了全球首个燕麦超级泛基因组和多组学整合数据库,突破了六倍体燕麦高效的基因组育种技术,实现了燕麦重要农艺性状相关基因的规模化挖掘与利用,并系统地揭示了燕麦环境适应性演化与表型可塑性的遗传学基础,相关成果发表在Nature Genetics、Nature Plants、Nature Communications、Molecular Plant、Cell Reports等国际知名期刊。
1)全球燕麦种质资源新质生产力的系统性评价与优异种质资源筛选
通过整合来自全球29个国家近万份燕麦种质资源,并结合地理来源多样性、表型多样性和部分遗传多样性等信息筛选了2300余份代表性燕麦材料,并于2022-2025年在河北保定、河北张家口、山东东营、青海等多个生态区域开展了连续4年的表型评价,完成了全球代表性燕麦种质资源新质生产力(基因型与表型)的全面调查,为了解我国现存燕麦种质资源的表型多样性、遗传多样性和育种潜力提供了重要参考(图1)。

图1张家口沽源县等多年多点的田间表型调查与杂交育种工作
同时,结合多年多点的表型调查与验证工作,筛选出了120余份高产、优质、耐盐碱、耐干旱的优异燕麦种质资源(图2),极大地丰富了我国优异燕麦骨干亲本库。

图2 高产、优质、耐逆燕麦种质资源挖掘与利用
2)全球首个燕麦多组学综合数据库助力燕麦分子辅助设计育种
结合全球1100余份野生和栽培燕麦种质资源的基因型图谱,多点多点的表型调查数据,不同发育时期和各类胁迫下多组学数据,以及通过全基因组关联分析和群体转录组分析获得的多个与产量、品质、抗病、耐逆等相关的候选eQTL和基因等信息,团队在国际重要期刊《Molecular Plant》上发表了全球首个燕麦多组学数据库的研究成果,公布了全球首个超过10 Gb的无间隙栽培六倍体燕麦参考基因组,并建立了OatOmics综合多组学数据库。该数据库整合了基因组、群体变异组、转录组、表型组等信息,为燕麦功能基因组学研究和分子育种提供了全面的共享平台(图3)(李伟等, Molecular Plant, 2025, IF=24.1)。OatOmics综合多组学数据库的建立标志着燕麦基因组学研究和分子育种进入了一个全新的时代,为科研人员和育种专家提供了高效、全面的数据平台,将极大地推动燕麦分子育种技术的发展。
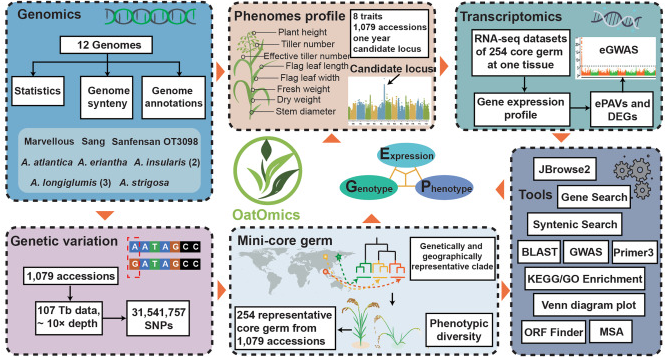
图3 OatOmics多组学综合数据库中数据概览
3)全球首个燕麦属超级泛基因组助力重要农艺性状相关基因的规模化挖掘与利用
燕麦属包含约30个野生近缘种,具有丰富的基因组倍型,展现出极高的表型多样性和广泛的适应性。然而,燕麦属各野生近缘种的起源与演化历程尚未明确,其蕴含的优异基因资源也未被充分挖掘,严重限制了野生近缘种在提升栽培燕麦抗逆性育种改良中的有效利用。2025年8月20日,团队在国际顶尖期刊《Nature Genetics》上公布了全球首个燕麦属超级泛基因组,利用35个高质量的燕麦基因组解析了燕麦属系统发育关系,对部分野生物种的分类提供了新的证据和见解,明确了燕麦属多倍体物种的起源与演化历史,揭示了燕麦属物种间复杂的网状进化和频繁的遗传交流塑造了其丰富的基因组倍型与基因组多样性。同时,在野生燕麦中挖掘到了约26.63%的新基因及59.93%的新基因型,并检测到栽培燕麦基因组中存在多个野生燕麦来源的大片段遗传渗入,证明了利用野生燕麦改良栽培燕麦的可行性,并凸显了其育种潜力(图4)(张宏宇等, Nature Genetics, 2025, IF=29.0)。这些研究不仅为解析燕麦进化与环境适应演化机制提供了理论支撑,也为挖掘与燕麦重要农艺性状相关的优异基因、推动燕麦分子设计育种以及高效利用野生资源改良栽培燕麦奠定了坚实基础。
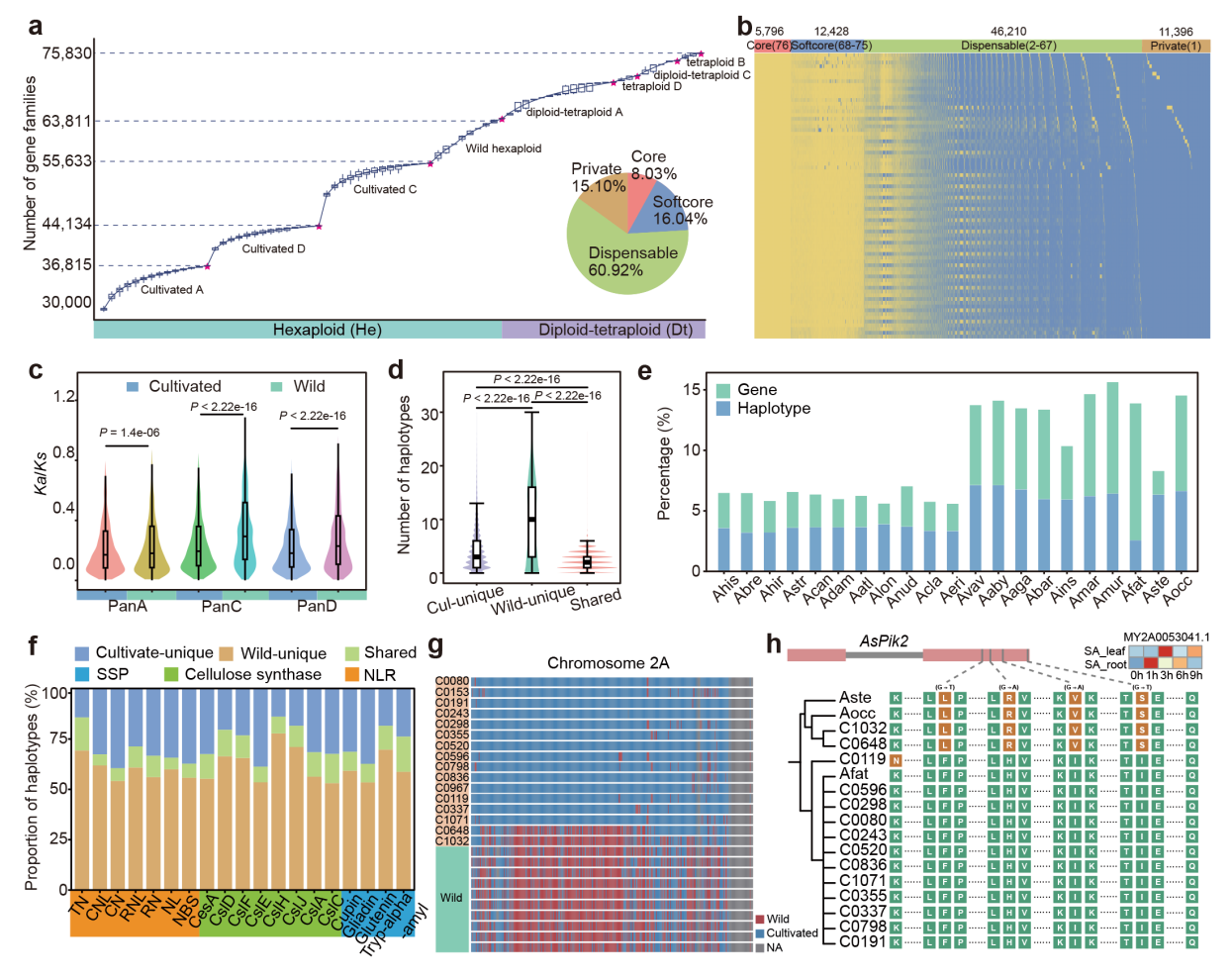
图4 燕麦属超级泛基因组构建及野生近缘种的遗传多样性
4)解码燕麦驯化轨迹与产量性状形成的遗传奥秘
栽培燕麦被认为是从六倍体野生祖先种不实野燕麦(Avena sterilis L.)驯化而来,该野生种具有极强地环境适应能力,蕴藏着大量可用于栽培种作物改良的珍贵遗传资源。然而,目前可获取的野生六倍体燕麦的基因组和遗传资源相对有限,这严重制约了对栽培燕麦的演化和驯化历史的认知,并限制了对影响燕麦重要农艺性状优异等位基因的探索。团队进一步通过解析不实野燕麦近乎完整的参考基因组,系统地揭示了野生和栽培燕麦的着丝粒序列结构全貌及动态演化历程,并鉴定到了燕麦早期驯化过程影响产量的关键基因组进化事件:栽培燕麦染色体4A和4D之间发现了一个约28 Mb的大片段复制,该片段携带多个与重要农艺性状相关的基因,并通过对全球117份野生和栽培燕麦材料的研究验证了这些基因在燕麦早期驯化中的重要作用(图5)(何强等, Nature Plants, 2024, IF=15.8)。该研究为揭示早期驯化对燕麦产量性状形成的遗传学基础研究和进一步改良栽培燕麦的产量性状提供了重要参考。

图5 燕麦早期驯化过程中影响产量的关键基因组进化事件
5)野燕麦超强环境适应性之谜被破译,为培育广谱适应性燕麦新品种提供重要指导
野燕麦 (Avena fatua L., 2n=6x=42, AACCDD) 因具有极强的环境适应性、胁迫抗性和入侵破坏性而被列为全球最具危害且分布范围最广的农田恶性杂草之一 , 对全球五十多个国家的粮食生产和生态环境造成了严重危害。尽管野燕麦对全球粮食安全造成了极大的威胁,但是因其丰富的种质资源,高度的表型可塑性,并能够在盐碱、干旱、除草剂、贫瘠等各类胁迫环境下仍然保持竞争优势,而被认为是栽培燕麦抗性能力提升和重要农艺性状遗传改良的天然宝贵资源 。然而,目前对于野燕麦的基因组学研究几乎处于空白状态,导致野燕麦具有强而广的环境适应性的遗传学基础尚不清楚,严重限制了野燕麦种质资源及优异基因信息的高效开发与利用。近日,团队结合野燕麦基因组学、群体遗传学和多组学等联合分析,系统地解析了野燕麦与栽培燕麦的遗传关系与演化历史;鉴定了野燕麦与栽培燕麦高度分化的基因组区域,证明了这些区域涵盖了大量与重金属、盐碱、干旱、高温、病虫害及除草剂等环境适应性和胁迫抗性相关的基因;挖掘到了参与野燕麦除草剂抗性的重要候选locus,并通过转基因等实验证明了4Dg0135144基因在燕麦除草剂抗性方面的重要作用(图6)(刘佳楠等, Nature Communications, 2025, IF=15.7)。该研究不仅为解析野燕麦环境适应性演化的遗传学基础提供了关键基因资源,同时也为培育高产、耐逆且具有广谱适应性的栽培燕麦新品种提供了重要指导。

图6 多组学解析除草剂耐受性关键基因及功能验证
6)揭示燕麦“皮”与“裸”的遗传密码,为皮裸燕麦种质的高效开发与利用提供重要指导
栽培燕麦根据成熟后的颖壳(外壳)是否紧密包裹籽粒,分为皮燕麦与裸燕麦两大类型。皮燕麦的籽粒被坚硬且高度木质化的颖壳紧密包裹,通常需脱壳处理后方可食用,适合机械收割及饲用。裸燕麦籽粒易脱落颖壳,具有高蛋白、高膳食纤维、低糖低脂等健康特性。但长期以来,皮燕麦与裸燕麦形态、营养、适应性等性状差异的遗传基础不清楚。2025年7月25日,团队结合431份全球燕麦种质资源的基因型图谱,解析了皮燕麦与裸燕麦的演化历史,揭示了结构变异在皮、裸燕麦种群分化、环境适应性和农艺性状形成中的关键作用,并鉴定了调控颖壳中木质素积累的重要候选基因(图7)(李伟等, Cell Reports, 2025, IF=6.9),该研究对于皮裸燕麦种质资源的高效利用以及燕麦品种的精准改良与分子设计育种具有重要意义。
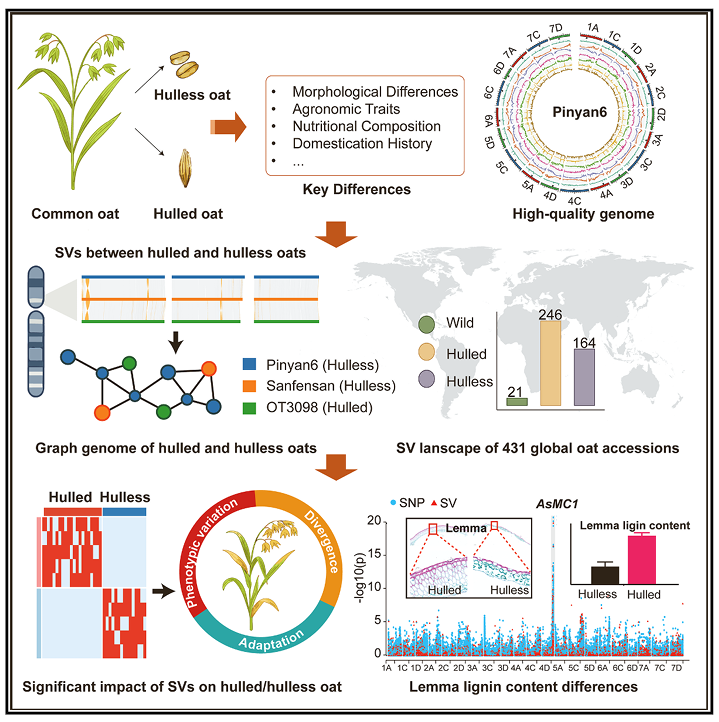
图7 燕麦有壳与裸壳分化的遗传密码
7)共表达网络助力燕麦抗旱基因挖掘与机制解析,填补燕麦功能基因组学研究空白
干旱是影响植物生长发育、限制作物产量最主要的环境胁迫之一,而植物抗旱反应的持续激活不利于其生长发育。相比于其他主要作物如小麦、水稻等,燕麦具有耐干旱、耐土地贫瘠和耐适度盐碱等特点,是挖掘新的抗旱优异基因和调控机制的重要遗传资源。然而燕麦在干旱胁迫下平衡其生长发育的分子调控机制仍不清楚。2025年3月9日,团队通过构建燕麦干旱胁迫和生长发育相关的共表达网络图谱,筛选了参与调控燕麦抗旱性与生长发育的关键候选基因,证明了AsHSFA2c正调控燕麦生长发育并负调控燕麦抗旱性的双重功能,并揭示了AsDOF25-AsHSFA2c-AsAGO1分子模块在调控燕麦生长发育与干旱胁迫响应平衡中的重要作用(图8)(刘宁坤等, Communications Biology, 2025, IF=5.1)。该研究为解析燕麦抗旱性的遗传学基础提供了理论依据,而且为抗旱高产燕麦新品种的分子育种提供了重要的基因资源。
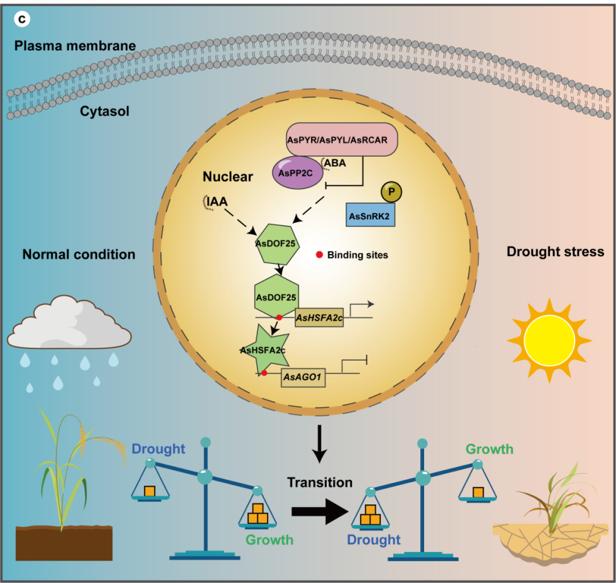
图8 AsDOF25-AsHSFA2c-AsAGO1分子模块调控燕麦抗旱性与生长发育的模式图
8)积极拓展合作网络,推动科研成果转化,构建“产学研”一体化的协作体系
紧扣国家基础研究与应用协同发展战略导向,团队积极构建“产学研”一体化合作体系,推动燕麦研究成果从实验室走向生产一线。目前团队已与多家中科院研究所、农科院研究所、高校、企业等建立了长期稳定的合作关系。如团队与张家口市农业科学院等单位签署正式战略合作协议(图9),聚焦品种选育、关键技术研发等核心领域开展联合攻关,助力区域燕麦产业提质升级;同时与国营企业-沽源牧场集团建立长期稳定合作关系,重点推进燕麦新品种示范推广、饲草高效利用技术集成等应用研究,将前沿科研成果深度融入生产实践。

图9 种质资源合作协议签署现场
参考文献:
1. Li, W., Wang, Y., Liu, J., He, Q., Zhou, Y., Li, M., Liu, N., Liang, H., Yun, Y., Gong, Z., et al. A gap-free complete genome assembly of oat and OatOmics, a multi-omics database. Molecular Plant, 2025, 18(2):179-182.
2. Zhang, H., Liu, N., Wang, Y., Zheng, X., Li, W., Liu, Z., Liu, J., Wang, Y., Xing, L., Li, T., et al. Super-pangenome analyses across 35 accessions of 23 Avena species highlight their complex evolutionary history and extensive genomic diversity. Nature Genetics, 2025, 57:2276-2288.
3. He, Q., Li, W., Miao, Y., Wang, Y., Liu, N., Liu, J., Li, T., Xiao, Y., Zhang, H., Wang, Y. R., et al. The near-complete genome assembly of hexaploid wild oat reveals its genome evolution and divergence with cultivated oats. Nature Plants, 2024, 10(12):2062-2078.
4. Liu, J., Liu, N., Yan, W., Hu, X., Wang, M., Qin, R., Ma, L., Li, W., Wang, Y., Sun, Q., et al. Reference genome and population genomic analyses reveal insight into herbicide tolerance in Avena fatua L. Nature Communications. 2025, 16(1):9851.
5. Li, W., Liu, J., Liu, N., Liang, H., Zhang, L., Sun, Q., Yan, W., Li, M., Liu, X., Yun, Y., et al. Genomic insights into the divergence between hulled and hulless oats. Cell Reports, 2025, 44(8):116055.
6. Liu, N., Li, W., Qin, Y., Yun, Y., Yan, J., Sun, Q., Du, C., He, Q., Wang, S., Gong, Z., et al. Comprehensive co-expression network reveals the fine-tuning of AsHSFA2c in balancing drought tolerance and growth in oat. Communications Biology, 2025, 8:393.
7. He, Q., Xiao, Y., Li, T., Wang, Y. R., Wang, Y. T., Wang, Y., Li, W., Liu, N., Gong, Z and Du H. High-quality genome of allotetraploid Avena barbata provides insights into the origin and evolution of B subgenome in Avena. Journal of Integrative Plant Biology, 2025, 67(6):1515-1532.